|
||||

Синантропные и домашние животные как резервуар коронавирусовУДК 578.42 Липилкина Т.А., Головин С.Н., Попов И.В., Липилкин П.В., Ермаков А.М. Глобализация и урбанизация являются процессами, которые опосредованно способствуют распространению различных инфекционных агентов среди людей через синантропных и домашних животных. Около 75% инфекционных заболеваний человека имеют зоонозный потенциал [1]. Геномы коронавирусов (CoVs) обладают значительной способностью к рекомбинации, и большинство CoV-инфекций человека являются зоонозными [2]. В большинстве случаев CoVs для первичного заражения человека необходимо преодолеть межвидовой барьер. Способность быстро приспосабливаться к новым хозяевам и экологическим нишам объясняется, прежде всего, высокой скоростью мутаций, имеющей причиной низкую надежность работы вирусной РНК-полимеразы [3]. Филогенетические исследования показывают, что летучие мыши являются резервуаром а- и в-коронавирусов, в то время как птицы -резервуаром у- и б-коронавирусов [4]. При этом а- и в—коронавирусы встречаются только у людей и млекопитающих, таких как собаки, кошки, свиньи, летучие мыши, мыши, крысы, лошади и крупный рогатый скот [5, 6, 7, 8], а у- и б-коронавирусы заражают в основном птиц, но, также, встречаются среди свиней [9]. Некоторые а- и в-коронавирусы вызывают у человека легкие (HCoV-229E, HCoV-NL63, HCoV-OC43 и HCoV-HKU1) и тяжелые острые респираторные синдромы (SARS, MERS и COVID-19) [10, 11, 12]. Несмотря на многие сомнения о природных резервуарах SARS и MERS, летучие мыши были определены в качестве наиболее вероятного промежуточного хозяина [13, 14]. При этом MERS-CoV имел самую низкую контагиозность среди людей по сравнению с SARS-CoV и COVID-19 [15, 16], так как не было зарегистрировано случаев передачи от человека к человеку. А штаммы HCoV-229E, HCoV-OC43, HCoV-NL63 и HCoV-HKU1 не вызывают системных воспалительных заболеваний у людей, что свидетельствует об адаптации этих штаммов CoVs к новым хозяевам [17, 18, 19]. При этом за всё время распространения SARS-CoV-2, как и другие CoVs, демонстрирует адаптивное преобладание контагиозности над вирулентностью [20]. Механизм взаимодействия CoV и хозяина основан на контакте его S-белка и АСЕ2-рецептора клеток у разных животных. S-белок состоит из двух субъединиц: S1 и S2. S1 взаимодействует с АСЕ2-рецептором человека с помощью Receptor Binding Domain (RBD) в области непосредственного контакта, называемой Receptor Binding Motif (RBM) [21]. S-белок является тем компонентом CoV, который, с большей вероятностью, может подвергнуться воздействию мутаций, в особенности тех, которые могут влиять на трансмиссивность [22]. Литературный обзор основан на поиске в Scopus, Medline/PubMed, Embase, Web of science за период с 2003 по 2021 годы. Поиск проводился по следующим комбинациям ключевых слов и SQL-оператора: «coronavirus» AND («animal» OR «bat» OR «rat» OR «cat» OR «dog» OR «mouse» OR «animal model» OR «stray animals»). Использована информация из 94 систематических анализов и исследовательских работ, обсуждающих фундаментальные и клинические аспекты CoV-инфекций у различных животных и их антропозоонозный потенциал. Коронавирусы летучих мышей. Летучие мыши могут жить относительно долго и летать, покрывая большие географические области, что позволяет CoVs распространяться среди них самих или переноситься к другим животным и людям [23]. Летучие мыши переносят CoVs без каких-либо клинических признаков, так как летучие мыши имеют физиологические и поведенческие особенности [24]. Например, высокая скорость метаболизма и, как следствие, высокая температура тела из-за полёта привели к изменениям [25], которые отразились на повышении толерантности иммунной системы и достижении эволюционного комменсализма с CoV [26, 27]. Постоянное увеличение контактов летучих мышей с другими животными играет ключевую роль в обширной межвидовой передаче CoV [28]. Толерантная иммунная система летучих мышей приводит не только к долгосрочному носительству [29, 30], но и рекомбинации, мутации и эволюции CoVs [31]. Таким образом, летучие мыши являются естественным резервуаром CoVs. Zheng-Li Shi и соавторы в своем исследовании обнаружили колонии летучих мышей-носителей штаммов RsSHC014 и Rs3367, у которых выявлено совпадение с геномом SARS-CoV на 85% и 96%, соответственно, а также был обнаружен штамм RaTG13 с геномом, идентичным SARS-CoV-2 на 96,2% [32, 33]. Возникающие в организме летучих мышей рекомбинации коронавирусов способствуют образованию новых штаммов, которые способны менять спектр хозяев и клеточный тропизм [34, 35, 36], а филогенетическое древо в-корона-вирусов показывает, что SARS-CoV-2 связан со штаммами CoVs летучих мышей Rhinolophus sinicus ZC45 и ZXC21, которые наиболее близки к штамму RaTG13. Исследовательская группа Zheng-Li Shi в 2014 году обнаружила у летучих мышей рода Pipistrellus штамм HKU5-CoV, у которого есть фу-риновый сайт для разрезания S-белка во время связывания с ACE2-рецептором. И ближайший родственник HKU5-CoV с похожим фури-новым сайтом — это SARS-CoV-2, который мог эволюционировать у таких летучих мышей, как Aselliscus stoliczkanus, Rhinolophus affinis и Rhinolophus sinicus [37]. После вспышки SARS в 2002 году распространение CoV среди людей было связано с мутациями в RBD S-белка, которые отличали эпидемический штамм от вирусов-предшественников, присутствующих у летучих мышей. Лабораторные исследования показали, что, хотя штамм HKU4-CoV является схожим с MERS-CoV, S-белок HKU4-CoV не способен поддерживать проникновение вируса в клетки человека до тех пор, пока не появятся необходимые мутации гена, кодирующего S-белок или иные молекулярные механизмы [38]. Немалое значение в процессе инфицирования имеет протеолитическое расщепление S-белка, а не только сам контакт S-белка с рецепторами человека ACE2 или DPP4. Именно протеолитическая активация ACE2-рецепто-ра с S-белком предполагает новый параметр для оценки потенциала возникновения CoV-инфекции у людей. Плазмин и другие протеазы человека могут способствовать расщеплению фуринового сайта в S-белке SARS-CoV-2 до его контакта с клеткой, что увеличивает кон-тагиозность и вирулентность [39]. CoVs с зоонозным потенциалом, подобные SARS-CoV-2, циркулирующие у летучих мышей в Юго-Восточной Азии, способны заражать клетки человека путем связывания с ACE2-рецептором без адаптации in vitro в первичной культуре клеток респираторного тракта человека [40, 41, 42], тогда как S-белки PDF-2180-CoV (MERS-Uganda) и HKU5-CoV способны связываться с клетками человека исключительно в присутствии трипсина. В экспериментальном заражении SARS-CoV-2 летучих мышей Rousettus aegyptiacus путём инокуляции, у испытуемых не было выявлено ни клинических признаков, ни летальных исходов. Кроме того, наблюдалось оральное и фекальное выделение CoV. Вирусная РНК была обнаружена с помощью полимеразной цепной реакции с обратной транскрипцией (ОТ-ПЦР) проб из эпителия дыхательных путей и пищеварительного тракта и связанных с ними лимфатических узлов. Аутопсия и гистологические исследования, проведенные на инфицированных летучих мышах, выявили эпителиальный некроз, отек и инфильтрации лейкоцитов в слизистой оболочке верхних дыхательных путей [43]. Это может свидетельствовать о попадании CoV во внешнюю среду с патологическими выделениями летучих мышей. Существует два основных механизма передачи CoV: контактный и аэрозольный [44]. Распространение SARS- и MERS-подобных CoVs в значительной степени происходит аэрогенно — в аэрозолях [45, 46, 47]. Поскольку время потери контагиозных свойств CoV в различных условиях и предметах окружающей среды до конца не изучено, то возможно рассматривать другие механизмы распространения. Исследования о конкретной эпидемиологической цепи передачи SARS-CoV-2 от резервуара к человеку свидетельствуют о том, что летучие мыши являются резервуаром SARS-CoV-2 и других CoVs, но прямая передача вируса от них человеку невозможна, либо возможна через промежуточного хозяина [48, 49, 50]. Было обнаружено, что пациенты разных регионов Азии инфицировались штаммами SARS-CoV-2 с различной вирулентностью [51]. Сравнительное исследование полных последовательностей генома 95 вариантов SARS-CoV-2, которые присутствовали в GenBank (NMDC) и хранилище генома NGDC (National Genomics Data Center), продемонстрировало связь между временем, местоположением сбора образцов и генетическим разнообразием. Обнаруженные мутации в штаммах SARS-CoV-2 позволяют предположить их разную эволюцию в разных географических местах, и это косвенно подтверждает распространение CoV летучими мышами и вероятность заражения людей [52, 53]. Однако S-белок SARS-CoV-2 малоэффективно связывается с АСЕ2-ре-цептором летучих мышей в исследованиях in silico и in vitro, что даёт возможность критично оценивать передачу SARS-CoV-2 человеку от летучих мышей [54, 55]. Необходимым условием передачи CoV является существование летучих мышей, как постоянной синантропной фауны с регулярными контактами с человеком. На первый взгляд — урбанизация способствует таким контактам, но есть ряд препятствующих факторов. Во-первых, необходимо учесть, что присутствие таких факторов городской среды, как шум, особенности ландшафта и уменьшение «зелёной среды» негативно отражаются на распространении колоний летучих мышей разных родов [56, 57, 58, 59]. Во-вторых, CoVs летучих мышей не более контагиозные [60], чем CoVs других животных. На сегодня нет убедительных доказательств того, что CoVs именно летучих мышей ухудшают эпидемическую обстановку в популяции людей [61]. Следует отметить, что из 54 разновидностей вирусов, которые переносятся летучими мышами, менее половины имеют потенциал передачи людям [62]. Отсутствие продромального периода и клинических симптомов в инфекционном процессе у летучих мышей от CoV снижает вероятность распространения инфекции. Также нельзя исключать, что антропогенная деятельность и употребление мяса летучих мышей, скорее всего, повлияли на появление пандемии COVID-19 не меньше, чем непосредственная жизнедеятельность летучих мышей [63]. Коронавирусы мышей и крыс. Вероятность передачи SARS-подобных CoV от мышей и крыс человеку является низкой, потому что у этих млекопитающих ACE2-рецепторы имеют чрезвычайно низкую аффинность к S-белкам SARS-CoV и SARS-CoV-2 [64]. В естественной среде CoV у мышей представлены вирусом мышиного гепатита (MHV) [65], а у крыс Rat-CoV: infectious bronchitis virus (IBV), который является общим и для кур. Также показана общая кластеризация CoV птиц с MHV A59 в исследовании Duraes-Carvalho и соавт. [66]. В исследовании Zhang и соавторы [67] проанализировали 177 образцов кишечника синантропных грызунов из трех различных видов (Apodemus chevrieri, Apodemus ilex и Eothenomys fidelis) и обнаружили РНК а- и в-коронавирусов в 23 образцах (13%). Мыши и крысы не являются потенциальными переносчиками CoV от резервуара к человеку, но трансгенные мышиные модели часто используются в экспериментах, связанных с исследованиями CoV [68, 69, 70, 71], и, тем самым, сохраняется вероятность техногенной вспышки. Более того, трансгенные мыши экспрессирующие человеческую 0026/ди-пептидилпептидазу 4 (DPP4) уже использовались при лабораторном заражении MERS-CoV, но в естественных условиях мыши не восприимчивы к этому CoV [72, 73]. Коронавирусы синантропных птиц. Были получены данные об увеличении популяции а-коронавирусов у разных видов птиц [74]. В общей сложности 500 образцов, включая трахеальные и клоакальные мазки, взятые у 312 диких птиц, были проанализированы с помощью ОТ-ПЦР. И 65 проб (13%) от 22 видов птиц были положительными на а-коронавирус. Молекулярно-эволюционный анализ показал, что нуклеотидные последовательности из образцов, собранных в Бразилии, не кластеризовались ни с одной из последовательностей генов а-ко-ронавирусов, хранящихся в базе данных GenBank. Птицы могут быть потенциальными новыми хозяевами, ответственными за распространение а-коронавирусов. При этом птицы летают на большие расстояния, что также потенцирует распространение CoV. В исследовании Zhuang и соавторы [75] проводили генетический анализ CoV у голубей и воробьёв, в том числе тех, которые используются в фермерской торговле, и пришли к выводу о массовой циркуляции среди исследуемых колоний птиц разных эволюционирующих штаммов CoV, не исключая возможности переноса CoV от голубей фермерским птицам. Также исследование 192 птиц на Ближнем востоке семейств: дро-финных, голубиных, соколинных, бекасовых, утиных, фламинговых, попугаевых и фазановых - выявило такие рекомбинации у б-корона-вирусов, которые обладают потенциалом межвидовой передачи млекопитающим, но б-коронавирусы были обнаружены лишь у 8 (4,16%) птиц с наибольшей относительной частотой штамма UAE-HKU30 у пе-репёлов (50%) [76]. Таким образом, синантропные птицы имеют потенциал, чтобы стать резервуаром патогенных для человека CoV, и, как переносчики, обладают всеми необходимыми качествами для поддержания персистенции и существования цепей передачи инфекции среди синантропной фауны и домашних животных. Коронавирусы собак и кошек. Коронавирус собак (CCoV) является представителем рода а-коронавирусов [77], а респираторный коронавирус собак (CRCoV) является представителем рода Бетакоронави-русов. CCoV имеет схожесть с вирусом трансмиссивного гастроэнтерита свиней (TGEV) и коронавирусом кошек (FeCoV); тогда как CRCoV связан с коронавирусом крупного рогатого скота (BCoV). На основании анализа генов, кодирующих M- и/или S-белок, штаммы CCoV были классифицированы на два генотипа: CCoV-I и CCoV-II [78, 79]. Также штаммы CCoV-II были дополнительно классифицированы на два подтипа: CCoV-IIa (классические штаммы) и CCoV-IIb (штаммы, возникшие в результате рекомбинации между CCoV-II и вирусом трансмиссивного гастроэнтерита свиней (TGEV)) [80]. Известно, что АСЕ2-рецепторы человека и собаки различны по 5 аминокислотным остаткам, что является преградой для эффективного прикрепления S-белка SARS-CoV-2 к АСЕ2-рецептору собаки. Однако в исследовании Sit и соавторы [81] были приведены случаи наличия бессимптомного клинического течения при заражении SARS-CoV-2 (при исследовании 15 собак из семей с подтвержденным COVID-19, положительный результат вирусологического теста был у 2 собак), что подтверждалось ОТ-ПЦР, секвенированием и иммуно-ферментным анализом (ИФА). При этом полностью исключается передача SARS-CoV-2 от одной собаки к другой, а наличие положительных результатов лабораторных исследований (ОТ-ПЦР и ИФА) при отсутствии реальных клинических симптомов у собак может являться ложноположительными результатами или свидетельствует о бессимптомной инфекции. Коронавирус кошек (FeCoV) встречается в двух серотипах, которые можно различить по их биологическому поведению, но не по морфологии патологических изменений, которые они вызывают. Оба серотипа способны вызывать инфекционный перитонит кошек (FIP). Серотип I более распространен в природе и отвечает за 80-90% естественных клинических случаев [82]. Серотип II встречается сравнительно редко, он возник в результате рекомбинации между FeCoV серотипа I и CCoV серотипа II после межвидовой передачи кошкам. Межвидовая передача CoV от собак кошкам, приводящая к генетической рекомбинации между серотипом I FECV и серотипом II CCoV, привела к появлению серотипа II FECV [83]. Гипотезы восприимчивости кошек к SARS-CoV-2 для контроля COVID-19 на сегодня не подтверждены. Jianzhong Shi и соавторы [84] делали попытку инокуляции кошкам SARS-CoV-2. Результаты исследования показали, что SARS-CoV-2 может реплицироваться у кошек. Подтвердилась экспериментальная аэрогенная передача SARS-CoV-2 от инокулированных кошек здоровым, хотя нет подтверждения такой передачи в естественных условиях. Напротив, есть подтверждения передачи SARS-CoV-2 больными COVID-19 людьми кошкам и собакам [85, 86, 87, 88, 89]. При этом есть данные, что кошки и собаки не имеют положительного результата ОТ-ПЦР и ИФА при контакте с заболевшими COVID-19 людьми [90, 91]. Но РНК SARS-CoV-2 обнаруживается в пробах со слизистой носа, ротовой полости, кошачьей шерсти и подстилке [92]. Предположительным фактором передачи CoV от бездомных собак и кошек являются их контакты с домашними питомцами и синантропными животными. Это благоприятно служит распространению CoV и создаёт постоянную эпизоотическую ситуацию неопределенного потенциала, но с формированием стационарных очагов инфекции.
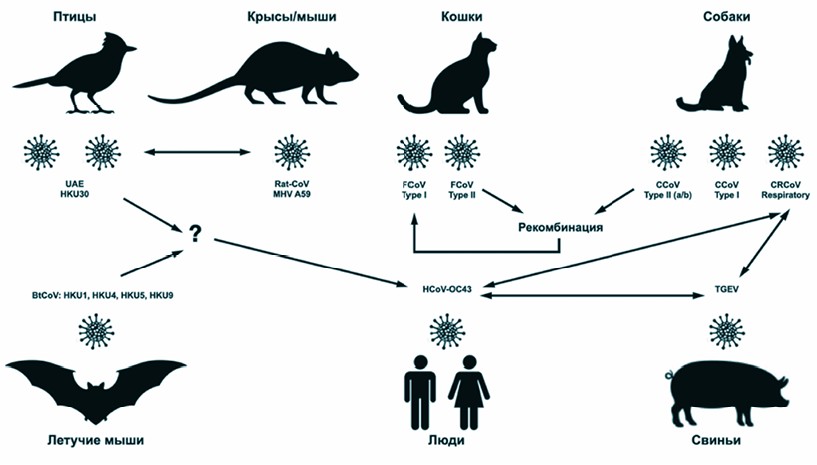
Рис. 1. Схема передачи коронавирусов от животных человеку на основе проанализированных данных литературы Выводы. Таким образом, в популяции некоторых синантропных летучих мышей и птиц регулярно присутствуют CoVs, которые могут пересечь видовой барьер с последующим заражением человека (рисунок 1). Преодоление CoVs межвидового барьера может сопровождаться как приобретением контагиозных свойств и увеличением вирулентности, так и их утратой и снижением вирулентности. В исследованиях не приведено подтверждающих данных о прямой передаче CoVs от синантропной фауны человеку. Кошки и собаки, в том числе и бездомные, достоверно не являются промежуточными хозяевами. Но антропогенное воздействие и урбанизация создают новые варианты взаимодействия птиц, летучих мышей, людей, бездомных кошек и собак, что не исключает эволюции CoVs и появления новых резервуаров инфекции в непосредственной близости проживания людей [93, 94]. Выявлены следующие облигатные факторы для переноса CoVs человеку: мигрирующая фауна с носительством разных штаммов CoVs и особенное антропогенное присутствие (мелиорация, субурбанизация). Данная работа выполнена в рамках гранта Российского фонда фундаментальных исследований № 20-04-60263. Список литературы:
Резюме. Заболевания, вызываемые коронавирусами, встречаются у животных и человека. Передача коронавирусов происходит как от человека к человеку, так от животного к человеку. При этом коронавирусам необходимо преодолеть межвидовой барьер для передачи человеку. Синантропные животные разные штаммов коронавирусов, некоторые из которых потенциально опасны для человека. Нельзя исключать, что антропогенная деятельность влияет на распространение коронавирусов среди людей не меньше, чем непосредственная жизнедеятельность животных в их ареалах. Летучие мыши являются резервуаром а- и в-коронавирусов, птицы являются резервуаром Y- и б-коронавирусов. Встречаются а- и в-коронавирусы среди людей и других млекопитающих, вызывая легкие и тяжелые острые респираторные синдромы. К тяжелым респираторным синдромам относят заболевания: SARS, MERS и COVID-19, возбудители которых вызвали эпидемии и пандемию среди населения в 2002, 2012 и 2020 годах. В популяции некоторых синантропных летучих мышей и птиц регулярно присутствуют коронавирусы, которые могут пересечь межвидовой барьер с последующим заражением человека. Немаловажную роль играют ареалы животных и их сезонное пересечение с местами проживания людей, а также особенности распространения коронавирусов во внешней среде в зависимости от физических условий: температура, влажность, движение потоков воздуха. Антропогенное воздействие и урбанизация создают новые «сценарии» взаимодействия людей и домашних животных с синантропными животными, что не исключает эволюции коронавирусов и появления новых резервуаров инфекции в непосредственной близости от проживания людей и их домашних животных. Авторами проведен анализ диапазона хозяев коронавирусов и вероятность их передачи от синантропных и домашних животных к человеку. Ключевые слова: коронавирусы, синантропные животные, летучие мыши, мыши, крысы, собаки, кошки, птицы, эпидемиология, эпизоотология. Сведения об авторах: Липилкина Татьяна Александровна, аспирант кафедры «Биология и общая патология» ФГБОУ ВО «Донской государственный технический университет»; 344010, г. Ростов-на-Дону, пл. Гагарина, 1; тел.: 8-905-4563847; e-mail: tterpitskaya@gmail.com. Головин Сергей Николаевич, ассистент кафедры «Биоинженерия» ФГБОУ ВО «Донской государственный технический университет»; 344010, г. Ростов-на-Дону, пл. Гагарина, 1; тел.: 8-918-5361591; e-mail: electromicrolab@gmail.com. Попов Игорь Витальевич, лаборант НИЛ «Центр агробиотехнологии» ФГБОУ ВО «Донской государственный технический университет»; 344010, г. Ростов-на-Дону, пл. Гагарина, 1; тел.: 8-999-6934028; e-mail: doc.igor.popov@gmail.com. Липилкин Павел Викторович, ассистент кафедры «Биология и общая патология» ФГБОУ ВО «Донской государственный технический университет»; 344010, г. Ростов-на-Дону, пл. Гагарина, 1; тел.: 8-918-5882467; e-mail: leeletter@ro.ru. Ответственный за переписку с редакцией: Ермаков Алексей Михайлович, профессор, доктор биологических наук, декан факультета «Биоинженерия и ветеринарная медицина» ФГБОУ ВО «Донской государственный технический университет»; 344010, г. Ростов-на-Дону, пл. Гагарина, 1; тел.: 8-928-2143344; e-mail: amermakov@yandex.ru.
|
| 2011 © Ветеринария Кубани | Разработка сайта - Интернет-Имидж | |
|---|---|---|